Ferramentas de diagnóstico xenético das abellas de mel europeas
2022/03/01 Gorrochategui, June - Applied Genomics and Bioinformatics taldeko ikertzailea (EHU) | Galartza, Egoitz - Applied Genomics and Bioinformatics taldeko ikertzailea (EHU) | Parejo, Melanie - Applied Genomics and Bioinformatics taldeko ikertzailea (EHU) | Estonba, Andone Iturria: Elhuyar aldizkaria

Apis mellifera ou abella de mel é na actualidade o polinizador máis importante do mundo, tanto ecolóxico como economicamente, e utilízase con frecuencia paira a produción de mel e a polinización de cultivos forestais. Con todo, estas abellas están ameazadas por diversos factores. Entre elas, a redución da diversidade xenética e a perda de subespecies locais adaptadas ao medio, teñen una notable influencia, tanto polo crecemento e importación comercial de raíñas a gran escala como pola apicultura migratoria a longa distancia. Paira garantir a saúde da poboación mundial é necesario asegurar a conservación das subespecies locais e manter a súa herdanza natural e a súa capacidade de adaptación. Europa é rica no patrimonio xenético das abellas de mel (representadas por polo menos 13 subespecies de 4 liñaxes evolutivas de 5 liñaxes definidas na especie) e foi una rexión fundamental paira a conservación e o desenvolvemento de ferramentas que a faciliten. Por iso, no marco do proxecto europeo SmartBees desenvolveuse un microchip de ADN que permitirá a identificación de subespecies de abellas de mel europeas. Diagnóstico.- Esta ferramenta facilitará e impulsará as actividades encamiñadas a preservar a conservación sustentable, o comercio controlado de raíñas, a verificación de produtos de apicultores e o patrimonio xenético das abellas locais.
Protexer a diversidade do mel
As abellas do mel (Apis melllifera) atópanse en diversos ecosistemas, distribuídos en varias lendas evolutivas e polo menos 30 subespecies. En Europa existe una gran parte desta diversidade, con numerosas subespecies endémicas divididas en catro liñaxes: África (A), Europa central e oriental (C), Europa occidental e setentrional (M) e Oriente Próximo e Asia Central (Ou). Con todo, a actividade humana ha ido diminuíndo progresivamente a diversidade xenética das abellas de mel europeas e o seu territorio natural. Precisamente polo comercio e importación de raíñas e a transhumancia de longa distancia, uno dos medos que existen é que as poboacións de abellas de mel autóctonas adaptadas ao medio veranse reducidas ou perdidas, representadas por abellas non autóctonas ou por simple hibridación. Está demostrado que as abellas de mel adaptadas ao medio teñen una maior capacidade de supervivencia. Por tanto, é importante fomentar o crecemento das abellas para que a abella poida durar a longo prazo.
En Europa puxéronse en marcha numerosos proxectos paira a conservación e o crecemento das abellas de mel autóctonas, entre eles os programas de mellora xenética. O éxito depende do control do campo de fertilización e do seguimento da orixe xenética da poboación. Neste sentido, é necesaria una ferramenta de análise xenética rápido, preciso e accesible.
O proxecto SmartBees comezou co obxectivo de desenvolver novas ferramentas moleculares que permitisen describir e conservar a diversidade das abellas de mel europeas. Así, mediante unha mostraxe exhaustiva da abella, deseñouse una ferramenta composta por marcadores xenómicos dun só nucleótido que permite describir a súa diversidade xenética (SNP) e identificar a subespecie de calquera abella europea.
Procedemento
Hanse muestreado 22 poboacións (Figura 1) que representan ás catro liñaxes evolutivos europeos e ás 14 subespecies que se poden atopar en Europa e rexións próximas. Paira a mostraxe das poboacións tomáronse 100 obreiras procedentes de colmeas non relacionadas entre si. En total, máis de 2.000 mostras, a mostraxe máis completa realizado até a data con abella de mel europeo.
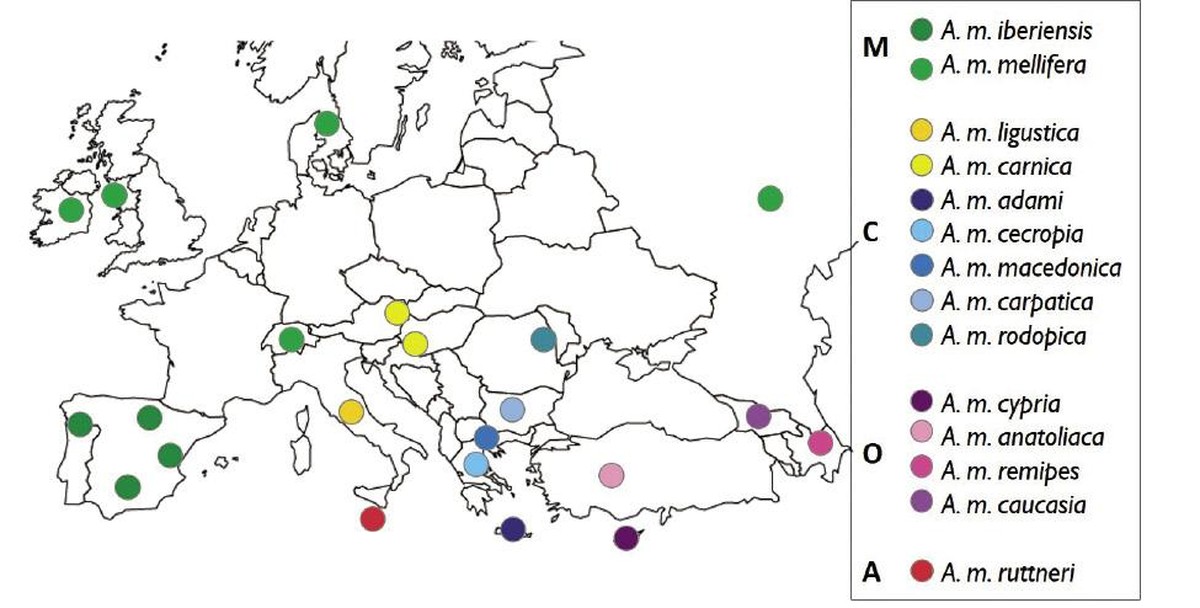
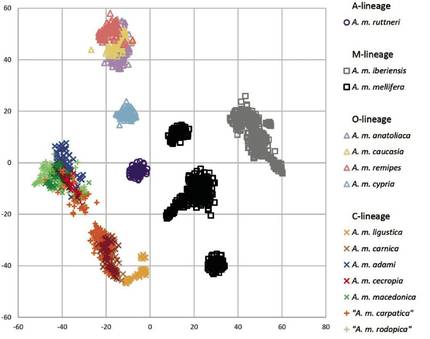
Una vez agrupadas as mostras segundo a poboación de orixe, extraeuse e secuenciado o seu ADN. A partir dos datos da secuenciación seleccionáronse os marcadores xenéticos con maior información sobre a orixe. É dicir, os marcadores xenéticos que mellor caracterizan ás 14 subespecies de abellas de mel de Europa, superando en total os 4.000 polimorfismos mononucleótidos (SNP). Os resultados da genotipación representáronse utilizando o diagrama t-SNE. Este diagrama agrupa as mostras da forma máis compacta posible nun mapa bidimensional, substituíndo cada individuo por un símbolo (Figura 2). Con este método, as abellas muestreadas agrupáronse en grupos illados (nubes de puntos) en función da súa liñaxe evolutiva ou subespecie. A única subespecie mostra da liñaxe africana, A. m. ruttneri, situouse no centro do gráfico, entre o resto de grupos. Na liñaxe Ou, as abellas A. m. cypria apareceron completamente separadas de tres abellas menos diferentes entre si (A. m. anatoliaca, A. m. caucasia e A. m. remipes). As dúas subespecies da liñaxe M foron ben diferenciadas, agrupando as poboacións de A. m. mellifera en tres subgrupos. Estes subconjuntos representan rexións de mostraxe remotas (rexión de Burzyan, Rusia, clúster supremo de A. m. mellife na figura 1) ou rexións illadas (Illa de Læsla, Dinamarca, cluster inferior de A. m. mellife). As mostras da liñaxe C dividíronse en tres subgrupos: (i) A abella A. m. ligustica, (ii) A. m. carnica xunto a varias abellas A. m. carpatica e (iii) un subgrupo heteroxéneo de abellas A. m. macedonica, A. m. cecropia, A. m. adami, A. m. rodopica e demais A. m. carpatica.
Ensaios adicionais e modelo de aprendizaxe automática
Ademais, hanse genotipado outras 1.900 abellas procedentes de colmeas paira a cría de SmartBees en toda Europa. Con estas 1.900 mostras, xunto coas 2.000 xa genotipadas (máis de 3.900 mostras en total), elaboraron un modelo estatístico que clasifica as abellas de mel europeas utilizando algoritmos de aprendizaxe automática. Este modelo permite calcular a probabilidade de que una mostra sexa una das 14 subespecies europeas. Os modelos automáticos de aprendizaxe teñen un lado bo, non se basean en hipóteses previas, polo que poden detectar diferenzas sutís. Esta característica foi especialmente importante paira o noso estudo, xa que nos permitiu diferenciar estas grandes cantidades de subespecies cunha estreita relación xenética. No artigo orixinal (Momeni et ao. 2021), pódense atopar máis detalles sobre métodos específicos de censura.
Precisión na asignación de subespecies
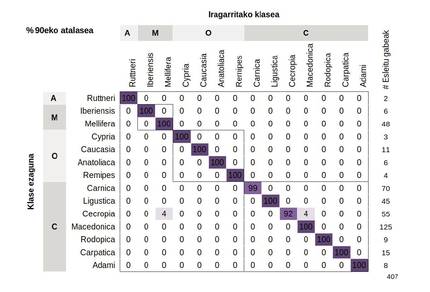
O modelo asignou correctamente a maioría das mostras cunha precisión media do 96,2%. A mellor forma de visualizar estes resultados é mediante unha matriz de confusión, que representa as porcentaxes de mostras previamente clasificadas correctamente ou incorrectamente (Figura 3). As cifras da diagonal central indican a porcentaxe de mostras correctamente clasificadas, mentres que os triángulos superior e inferior representan as mostras mal clasificadas. A clasificación será errónea cando as mostras anunciadas polo modelo e as etiquetadas sexan subespecies diferentes. Estes erros producíronse, por unha banda, cando as etiquetaxes das mostras ensaiadas foron erróneos e, por outro, cando as diferenzas entre as poboacións de referencia foron mínimas, ben pola súa proximidade xeográfica, ben por interferencias humanas.
Para que este modelo sexa aplicado nos programas e programas de conservación establecemos un limiar mínimo do 90%. Así, se o prognóstico dunha mostra é inferior ao 90%, clasificarase como “non asignado”. Se supera o límite asignarase á subespecie correspondente.
Retos da diversidade e próximos pasos
A diversidade das abellas de mel europeas supuxo un gran reto á hora de deseñar una ferramenta de diagnóstico de subespecies. A diverxencia das liñaxes evolutivas ha feito posible que a diferenciación xenética sexa sinxela, utilizando uns poucos SNP. Con todo, a distinción entre subespecies foi máis difícil debido a que a diverxencia entre especies do mesmo soberano foi recente e, por tanto, xeneticamente similares. Ademais, nalgunhas rexións europeas non están definidas as fluctuaciones do cambio de subespecies A. mellifera, e a entrada artificial de abellas estranxeiras ha difuminado as fronteiras naturais entre subespecies. Tamén pode ocorrer que os programas nacionais haxan interferido no fluxo natural dos xenes e alterado o patrimonio xenético da subespecie orixinal. En consecuencia, algunhas subespecies clasificáronse facilmente mediante o noso instrumento, mentres que outras mostras quedaron sen asignar. Con todo, trátase dunha ferramenta dinámica adaptable que permite mellorar a base de datos de referencia e/ou introducir novas subespecies. Neste sentido, tendo en conta as investigacións en curso, o método é aplicable á siciliana A. m. de Sicilia.
Dispoñibilidade
Con este instrumento poderase determinar a orixe xenética de máis mostras, o que resultará imprescindible en moitos aspectos: os apicultores poderán determinar o seu subespecie e grao de hibridación de abellas e dar aval aos seus produtos; os responsables da conservación europea poderán controlar as taxas de hibridación de colmeas en depósitos; os veterinarios poderán controlar o comercio de raíñas; e os criadores das abellas asegurarán a súa subespecie de raíñas.
Publicación científica completa
Agradecementos
Esta investigación levámola a cabo con mostras de apicultores, criadores e colaboradores. Agradecemos a súa colaboración. O proxecto SmartBees foi financiado pola Comisión Europea dentro do programa FP 7 KBBE (02.01.2013, Grant nr. 613960). Melanie Parello recibiu subvención do Goberno Vasco (IT1233-19).

Gai honi buruzko eduki gehiago
Elhuyarrek garatutako teknologia






