Libérer le noeud des maladies génétiques
2017/03/01 Garcia Etxebarria, Koldo - Genetikan doktoreaEHUko Genetika, Antropologia Fisikoa eta Animalia Fisiologia saila Iturria: Elhuyar aldizkaria

La base de nombreuses maladies, environ 14.000, se trouve dans les défauts génomiques. Ces maladies peuvent être classées en deux grands groupes : d'une part, lorsqu'un gène unique le provoque, elles sont appelées maladies monogéniques ou mendéliennes ; d'autre part, lorsqu'interviennent plusieurs gènes, appelés maladies génétiques complexes. La première étape est de découvrir quels sont les gènes liés aux maladies, qui est indispensable pour trouver remède. Même si ces dernières années, il ya eu de grands progrès dans la connaissance de ces gènes défectueux, la solution n'est pas si claire.
Les maladies causées par les gènes uniques, environ 4000, ont été les premières à être analysées. En suivant les modèles qui prédisent les lois de Mendel, il est plus facile de trouver un gène lié à la maladie. Pour ce faire, on analyse la distribution de la maladie dans une famille et on la classe en fonction de celle-ci.
Lorsqu'il est produit par un gène qui se trouve dans l'un des 22 chromosomes non sexuels, il est appelé une maladie autosomique. Dans les autosomiques dominants, la seule copie défectueuse du gène est suffisante pour développer la maladie. Parmi elles, les plus fréquentes sont l'hypercholestérolémie familiale (un sur 500 nouveau-nés), la neurofibromatose de type I (un sur 2.500) ou la maladie de Huntington (un sur 15.000).
Dans les maladies autosomiques récessives, les deux copies du gène doivent être défectueuses pour le développement de la maladie. L'anémie falciforme (une sur 625), la fibrose kystique (une sur 2.000) et la maladie de Tay-Sachs (une sur 3.000) sont les maladies les plus répandues de ce type.
En ce qui concerne les chromosomes sexuels, parmi les maladies liées aux gènes situés dans le chromosome X se trouvent la dystrophie musculaire de Duchenne (une sur 7.000) et l'hémophilie (une sur 10.000). Dans le cas du chromosome Y, cependant, ils sont très rares et provoquent généralement la stérilité.
Bien que nous ayons donné l'exemple des maladies monogéniques les plus répandues, la plupart des maladies mendéliennes sont des maladies rares, soit moins d'une personne sur 2000 en souffre. Cependant, le gène responsable est connu. Lorsqu'on soupçonne un patient de souffrir d'une maladie, on peut séquencer ce gène concret et confirmer s'il a cette mutation connue (changement de séquence). Cependant, il peut arriver qu'il n'y ait pas de mutation connue, c'est-à-dire que ce patient a une nouvelle mutation. Pour rechercher cette nouvelle mutation, les techniques de séquençage de tout le génome qui ont été développées ces dernières années sont très utiles.
Actuellement, nous sommes en mesure d'obtenir la séquence de la région fonctionnelle de tous les gènes (environ 20.000) d'une personne (ce qui est connu comme exome) et d'analyser les mutations qu'ils portent. Il est courant qu'il y ait certains changements, puisque le matériel génétique de tous les êtres humains n'est pas exactement le même ; en outre, la plupart des changements n'interfèrent pas dans la fonction du gène. Mais en comparant l'exome du malade avec celui de ses parents on peut déduire quels changements il a reçu de ses parents et quels sont nouveaux, et lesquels d'entre eux peuvent avoir des effets défavorables sur la fonction du gène.
D'autre part, les maladies génétiques complexes sont plus répandues dans la population, mais il n'est pas si facile de trouver les gènes responsables de celles-ci. Des maladies comme l'asthme, la sclérose en plaques ou le diabète. En outre, malgré sa composante génétique, en raison de l'influence du milieu, l'étude de la maladie est plus compliquée.
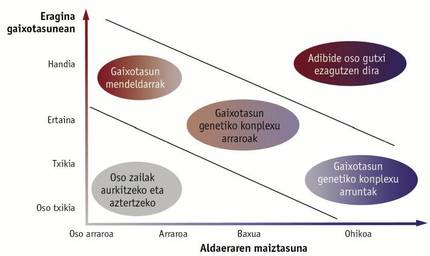
Pour identifier les gènes impliqués, la technique appelée analyse associative de l'ensemble du génome a été utilisée. Cette technique analyse les marqueurs génétiques SNP (de l'anglais single nucléotide polymorphism, polymorphisme mononucléotide) dispersés dans tout le génome. Des outils ont été développés pour analyser simultanément des milliers de marqueurs de ce type, appelés microarray, ce qui permet de connaître de manière simple et rapide la variété de SNP que chaque personne analysée porte. Si une variante de SNP est plus fréquente chez les personnes atteintes de maladie que chez les personnes saines, on peut soupçonner que cette variante est liée à la maladie. Près de 2 400 études de ce type ont été réalisées, qui ont associé de manière significative près de 17 000 SNP à une maladie ou caractéristique complexe (figure 1).
Cependant, nous ne sommes pas en mesure d'identifier tous les gènes associés à des maladies génétiques complexes. Nous savons qu'il y a peu de gènes qui influencent la maladie, mais ceux-ci ne sont pas suffisants pour expliquer complètement le développement de la maladie. De nombreuses maladies génétiques complexes sont dues à la combinaison de nombreux SNP communs, qui augmentent ou réduisent légèrement le risque de maladie (Figure 2). De plus, ce groupe de SNP n'a pas besoin d'être dans un gène, c'est-à-dire qu'il n'a pas besoin de créer une erreur dans un gène. Cela rend difficile l'interprétation qu'un ou plusieurs SNP sont liés à une maladie. Il se peut que ces SNP soient proches d'une mutation qui provoque vraiment une erreur et n'ont pas d'effet réel. Il peut également arriver que le SNP influence la régulation du gène et que, même si le gène ne présente pas de défauts, il ne soit pas à la hauteur nécessaire. Il reste encore beaucoup à faire pour connaître l'origine génétique de maladies génétiques complexes, et tout cela sera un défi pour l'avenir immédiat.
Une fois que nous serons en mesure d'établir l'origine d'une maladie génétique, le défi suivant serait de trouver votre remède. Jusqu'à présent, les seules solutions réussies ont été l'atténuation des effets de la maladie ou la prévention des conditions environnementales qui peuvent la produire. En fait, la réparation directe des défauts géniques, la thérapie génétique, n'a pas été aussi réussie que nous le voulions jusqu'à présent, de sorte qu'il n'a pas été généralisé.
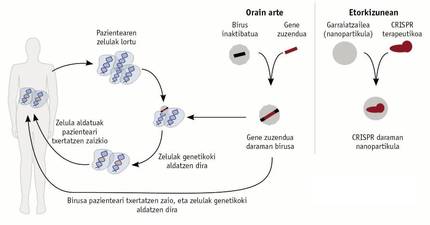
Dans les cas où un gène donné n'est pas en mesure de remplir sa fonction, des essais ont été effectués pour incorporer un gène qui fonctionne correctement dans le matériel génétique de certains patients (figure 3). Pour ce faire, un gène fonctionnel est introduit dans un virus modifié, qui est administré au patient comme traitement et intégré dans le matériel génétique des cellules du patient. Cette stratégie a commencé à être appliquée chez certains patients avec une immunodéficience grave combinée en 1990, mais son efficacité était à court terme. À la recherche d'une solution à long terme, la technique a été améliorée et de nouveaux essais ont été effectués en 2000. Malheureusement, comme conséquence marginale de ces nouveaux essais, les patients ont développé une leucémie qui a provoqué leur abandon. Cet effet collatéral avait pour origine que le virus introduisait la copie fonctionnelle du gène dans un autre gène, ce qui générait de la leucémie.
Depuis 2015, il semble que nous vivions une résurrection des thérapies géniques. Des techniques comme le CRISPR, les doigts de zinc sont capables d'éviter les limitations et les problèmes associés à l'utilisation de virus (figure 3). Ces techniques sont capables de connaître le gène que vous voulez résoudre, de sorte qu'ils ont une spécificité que les virus n'ont pas. Ces techniques donnent déjà beaucoup à dire et à court terme vont donner encore plus. Leur efficacité doit être vérifiée.
Le développement d'outils pour connaître les bases des maladies génétiques nous a permis de mieux comprendre ces maladies et il semble que nous avons commencé à imaginer des remèdes efficaces. Bien que prudemment, nous pouvons être optimistes et penser que dans un proche avenir nous allons vivre une révolution autour des maladies génétiques.
Bibliographie Bibliographie

Gai honi buruzko eduki gehiago
Elhuyarrek garatutako teknologia