Descodificado el genoma del microorganismo responsable de la malaria
2002/10/03 Roa Zubia, Guillermo - Elhuyar Zientzia
Ahora, tres grupos de bioquímicos han desarrollado un proyecto para descodificar el genoma del microorganismo P. falciparum. Un equipo pertenece al laboratorio inglés Sanger Centre (Cambridge) y ha decodificado nueve cromosomas. El resto de los cromosomas han sido descodificados por dos grupos de Estados Unidos, del Instituto TBAJO (Maryland) y de la Universidad de Stanford (California).

Paralelamente, se ha descodificado la de otro microorganismo similar, el Plasmodiun yoelii yoelii, responsable de la malaria en las ratas; comparados ambos, los científicos sabrán qué hace que un microorganismo sea perjudicial para el ser humano y que el otro no lo sea.
Los resultados de estos estudios se han publicado en el número de esta semana de la revista Nature. Al mismo tiempo se ha descodificado el genoma del mosquito Anopheles gambiae, que transmite la malaria. La investigación de Eltxoa la ha dado la revista Science. Para destacar la importancia de los trabajos realizados, los responsables de las investigaciones y de las dos revistas ofrecieron ayer unas ruedas de prensa en Washington y Londres. Al igual que se hizo con el proyecto Genoma Humano, han decidido publicar todos los resultados al mismo tiempo y han anunciado con gran fuerza.

Genoma pequeño
Los científicos han dedicado mucho tiempo a este trabajo, algo muy significativo. Puede pensarse que descodificar completamente el genoma de cualquier organismo es y es así, pero teniendo en cuenta el tamaño de este genoma ha sido demasiado tiempo.
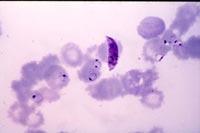
P está formado por 23 millones de pares de bases. ADN del Falciparum, dividido en 14 cromosomas. Los datos no son más que números, aunque, por ejemplo, el genoma de la mosca Drosophila melanogaster, con 120 millones de pares de bases, fue descodificado en menos de un año. Sin embargo, el proyecto de genoma de este microorganismo se puso en marcha en 1996, y es una consecuencia clara de las dificultades que se han encontrado a lo largo del tiempo.
El origen de estas dificultades radica en los componentes del genoma, en realidad la metodología que ha sido exitosa con otros genomas no es apropiada para el estudio del Plasmodium falciparum. Aunque las bases que utiliza el ADN son cuatro, G, C, T y A, este microorganismo tiene secuencias muy largas compuestas únicamente por las bases A y T. Por tanto, para descodificar el genoma, el ADN se rompe en pequeñas partes, después de descodificar cada trozo, se ordena informáticamente para buscar la secuencia original. Este último paso se complica cuando en lugar de cuatro bases sólo participan dos.
En las estructuras cromosómicas de este microorganismo se han encontrado también una serie de peculiares características. Por ejemplo, la estructura de los telómeros extremos es muy compleja, lo que, según los científicos, facilita el paso de mutaciones, muchas de ellas se producen en esas zonas del genoma. Por tanto, los Plasmodium son microorganismos bastante variables, lo que impide avanzar en la investigación de la malaria.
El misterio de los metabolitos
Además de las dificultades de descodificación, estas secuencias generan problemas de interpretación. Nosotros sabemos que este tipo de secuencias no codifican género, pero puede que haya que reconsiderarlos.
Durante la investigación se ha utilizado software que detecta dónde se encuentran los genes (con intrón), pero estos programas no son exactos y sus criterios para identificar un gen son insuficientes o correctos. En el caso del genoma humano también existía el mismo problema, ya que todavía no es posible decir cuántos genes tiene el ser humano.
La estrategia que se utiliza en la actualidad es la comparativa con genes conocidos. Para ello se utilizan genes de otros organismos similares. Por supuesto, esto no es un éxito, ya que se encuentra con las características genéticas propias de cada especie. El genoma del Plasmodium falciparum, además, ha provocado grandes sorpresas. Por ejemplo, las proteínas típicas que "gestionan" las moléculas básicas que transportan energía no han sido expuestas en estos estudios, ni de la ATP ni de la NADH.
Además, se han encontrado pocas proteínas de transporte. Sin embargo, los investigadores han detectado al menos una señal de la actividad mitocondrial y han identificado muchos genes relacionados con los apicoplastos (zonas de síntesis de ácidos grasos).
¿Qué significa todo esto? ¿Estamos ante un nuevo tipo de metabolismo? ¿O la metodología para encontrar proteínas todavía es muy retrasada?
¿Nuevos medicamentos?
La no comprensión del metabolismo del Plasmodium falciparum no significa que no puedan diseñarse nuevas estrategias contra él. De hecho, se han identificado cinco nuevas proteínas que participan en vacuolas de alimentación en esta vía y que podrían ser bloqueadas mediante inhibidores específicos.
Es más, el comportamiento exacto de la quinina y sulfanilamida que se usaban contra la malaria es evidente desde hace tiempo. Por lo tanto, hay una gran esperanza. En este camino también puede ser de gran ayuda la descodificación del genoma del mosquito Anopheles gambiae. Pero los científicos que han participado en el proyecto han trabajado con otra duda: ¿cuál es la mejor manera de combatir la malaria? ¿Proyectos genéticos de alto nivel? ¿Programas convencionales de vigilancia de la salud pública? La solución puede ser utilizar ambas vías simultáneamente. Los que ya han descodificado el Plasmodium falciparum han comenzado a descodificar los genomas de otros microorganismos. Habrá que esperar a ver hasta dónde llegan los resultados de este tipo de investigaciones.
Artículos complementarios:
Malaria o paludismo, el genoma de la mosquita que transmite
Malaria para un terrible problema de salud

Gai honi buruzko eduki gehiago
Elhuyarrek garatutako teknologia






